在高通量檢測這個領域里,基因芯片和二代測序這對相愛相殺的cp,原本均是了解基因組結構和功能的絕佳高手,如今為了一爭高下,又是鬧的不開交。這不,基因芯片仗著自己老大哥的身份,手持一個二維的DNA探針陣列所形成的三維地圖,以數以萬計的探針做方向標,能按圖索驥的找到基因組的特定位置,且結合完整成熟的指控分析手段能快狠準地篩選出所需基因,頗有幾分笑傲江湖的氣勢。
然而,RNA測序技術作為后起之秀,不僅能輕而易舉地解讀由轉錄組組成的生命雜志,就連探索新的突變位點以及新基因之事也都不在話下。這鋒芒畢露的架勢,顯然來勢洶洶,眼瞅著就要把這些年略顯沉寂的芯片老大哥給打壓下去,成為高通量領域里獨霸一方的勢力了。

至此,進入看戲模式的吃瓜群眾—科研者們,雖然表面看的熱鬧,其實內心深處也頗為糾結,萬一哪天真要戰隊,我該支持誰呢?芯片與測序各有所長,均可大放異彩,到底誰才是笑到最后的那一個呢?
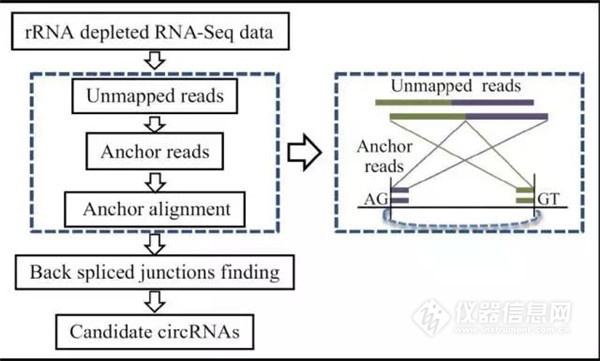
芯片vs測序,各領風騷
BMC Genomics 的一篇文章,似乎能為一眾心有存疑的科研者們指點迷津。它以9對肺鱗狀細胞癌配對樣本為材料,分別用Affymetrix HumanTranscriptome Arrays 2.0 (HTA)(最新一代全轉錄本表達譜芯片)和IlluminaHiSeq 2000檢測平臺,以測序200 M reads為基礎,深入比較了芯片和測序在轉錄本定量檢測和可變剪切方面的情況。
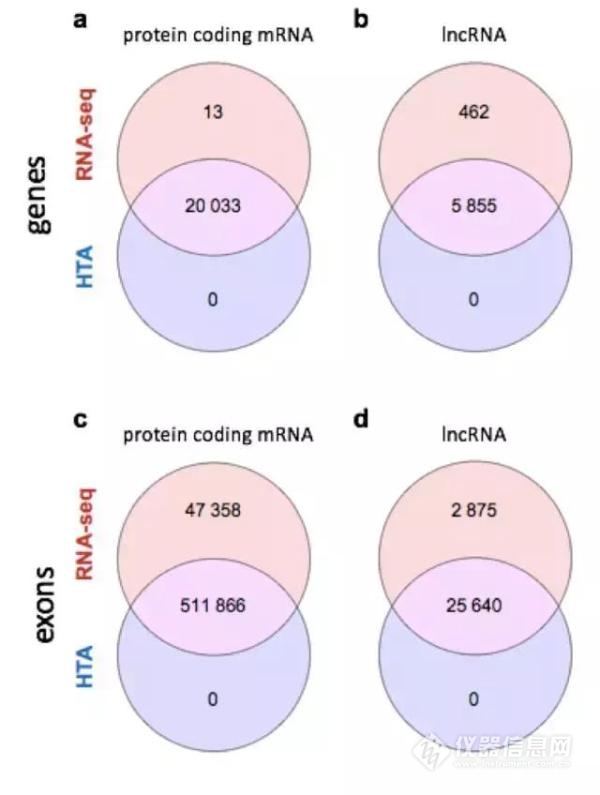
首先,就檢測基因覆蓋范圍而言,芯片和測序均可檢測到Ensembl數據庫中92%的mRNA和90%的lncRNA,可以說是不相上下,打成平局。
而在正常和腫瘤組織生物學重復比較分析,發現在低表達基因上,RNA-seq檢測的樣本可變性更大,而芯片則沒有表達高低的偏好性,說明HTA芯片的穩定性比RNA-Seq更好。
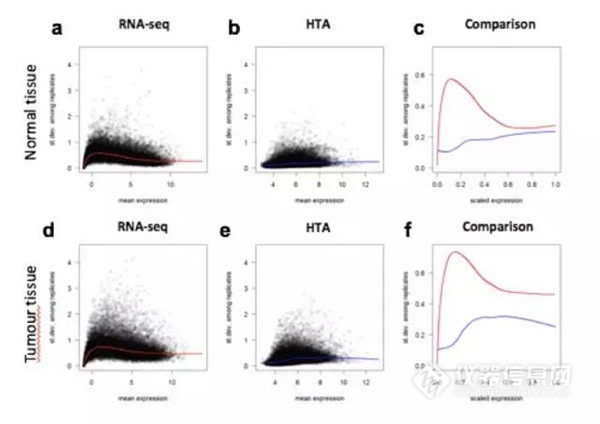
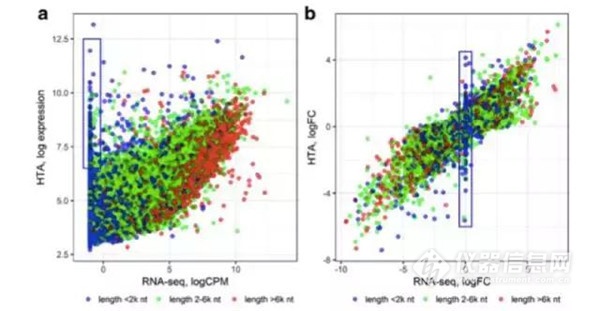
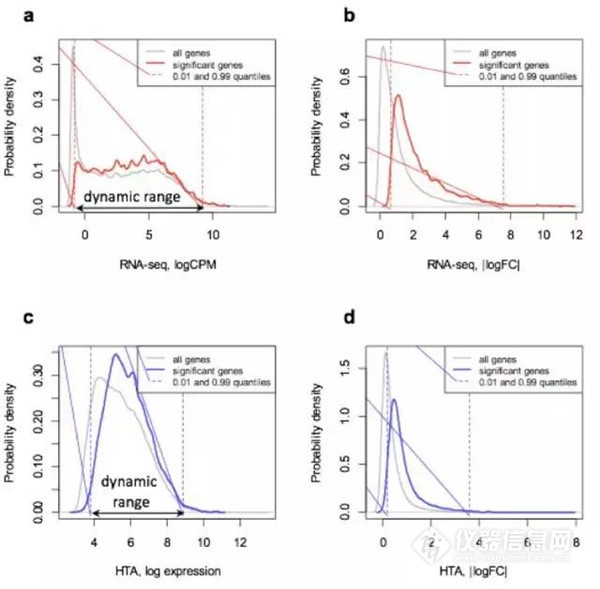
至于靈敏度,依據檢測信號分布強度(log2 intensity values),HTA的變化范圍在3到13之間,而RNA-seq則在-1到14之間,其動態范圍更大,可減少一些漏網之魚,然而對于一些短片度基因,卻是芯片的檢測效果更好一些。
而非配對差異分析結果中,在mRNA差異檢出數量中,HTA對比RNA-seq分別是6173和4777(3683為共有);在lncRNA差異檢出數量中,HTA對比RNA-seq分別是1219和892(376為共有)。
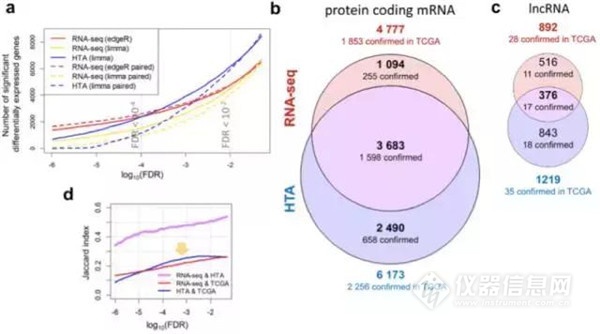
顯然,對同樣疾病樣本在TCGA上的數據分析比較,差異基因中,無論是mRNA,還是lncRNA,芯片與TCGA吻合的基因數量更多。