本文介紹了使用Thermo Scientific公司的TSQ Quantum Ultra質譜儀進行復雜生物機體中目標肽的方法,結果表明,基于串連質譜的選擇反應監測(SRM)流程,該方法可以在具有高化合物背景的復雜基體中檢測低濃度水平肽,同時保證良好的定量準確度和精密度。
目前一般生物標志物實驗的終點是發現公認的標志性蛋白名單,接下來的一步就應該是在擴展患者群體內完成對這些靶向蛋白的定量測定,以評價它們作為標志物的有效性。對這些在復雜基體中的靶向蛋白進行可靠的定量分析,其分析準確度和精密度是必需的。對低豐度蛋白質進行高通量檢測,對于寬動態范圍、高靈敏度和穩健易用性都有嚴格的要求。基于串連質譜的選擇反應監測(SRM)流程,對于復雜生物基體中所研究蛋白的特異肽可以進行高靈敏度和高選擇性的定量,同時也可以獨立進行生物標志物驗證[1-3]。該方法的主要優點是具有很低的檢測限,可以在具有高化合物背景的復雜基體中檢測低濃度水平肽,同時保證良好的定量準確度和精密度。
本文驗證了TSQ Quantum Ultra質譜儀在用于人血清酶解液中待測肽的定量分析時,顯示出高選擇性和高靈敏度的優勢。研究建立了通過高分辨率的母離子分離和目標肽多碎片離子時間序列檢測,同時進行目標肽的鑒定和定量的測定方法,對H-SRM方法的定量準確度也進行了評價。
實驗條件樣品制備
25μl人血清(Sigma Corp., St. Louis,MO)用975μl 濃度為100mM的碳酸氫銨緩沖液40倍稀釋,超濾處理(截留分子量10 000 MW),酶消化。將消化液真空干燥,并在0.1%甲酸250μl(10倍稀釋)水溶液里重組。
液相條件
納流高效液相(Nano-HPLC)
Thermo Scientific SurveyorTM MS 泵和MicroAS自動進樣器
色譜柱:75μm×100 mm C18
Post-split流速: 300nl/min
流動相: A為0.1% 甲酸的2%乙腈溶液;B為0.1% 甲酸的乙腈溶液
線性梯度: 90min,2%B升為50%B
進樣量: 2μl,直接進樣色譜柱
質譜條件
TSQ Quantum Ultra質譜儀
Thermo Scientific Ion MaxTM離子源,配有納流柱接頭(New Objective)
高選擇性反應監測(H-SRM):Q1,0.2FWHM;Q3,0.7FWHM
選擇反應監測(SRM):Q1,0.7 FWHM;Q3,0.7FWHM;Q2,1.5mTorr
定量增強數據關聯掃描(QED-MS/MS):(1)選擇反應監測掃描(SRM)為Q1和Q3,0.7FWHM;Q2,1.5mTorr;318種躍遷H-SRM實驗中駐留時間為5ms,循環時間2.2s。(2)定量增強數據關聯掃描(QED-MS/MS): 根據掃描1的母離子關聯掃描;信號閥值 5000;Q1和Q3,0.7FWHM;Q2,1.5mTorr;CE, 0.034×母體質量m/z + 3.134;動態排除,重復2次;持續時間,60s;排除時間,60s;排除名單大小,50;循環時間3.84s。
數據庫檢索
為方便檢測結果比較,所有的數據采用MascotTM數據軟件(Matrix Sciences)處理,使用SwissProt數據庫(分類:人類)。
肽選擇和SRM離子對參數設置
有兩種進行肽選擇和SRM離子對參數設置的基本方法[3]。如果靶向蛋白曾經在以前的LC-MS/MS實驗中被檢測過,該蛋白產生的特異肽就被選出,用于隨后的SRM實驗。相反,如果沒有LC-MS/MS數據可以用于靶向蛋白,肽選擇和SRM實驗設置則使用已知的蛋白序列(模擬方法)在電腦中完成。我們開發了一個用于靶向蛋白定量的SRM工作軟件,它可以用于預測候選肽,并可以為SRM選擇多碎片離子,從而建立儀器方法和序列文件,并可自動進行肽鑒定和定量數據處理(見圖1)。本文所述所有SRM實驗都使用了該軟件。
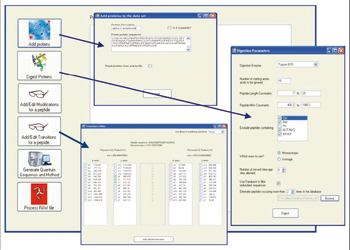
圖1. SRM工作軟件中肽選擇和SRM離子對參數設置。
結果和討論
H-SRM目標肽定量方法評價
本文所提出的H-SRM工作流程如圖2所示。高分辨分離(0.2FWHM)的主要優勢是它從復雜基體中選擇性檢測低豐度肽峰的能力3。通過監測在同一持續時間內選擇的多種碎片離子,完成對肽的鑒定。該方法沒有限定對每種肽可以選擇的碎片離子的數目。一般而言,每種肽選擇4~7種y-型離子足以保證實驗的高度專一性。對高分辨母離子分離的增強選擇性避免了費時的全掃描MS/MS。采用H-SRM (318組 H-SRM實驗為2.2s,駐留時間設為5ms)縮短了工作循環時間,使得在一個LC流程內不僅可以同時進行肽的鑒定和定量,而且還最大程度保證了檢測靈敏度。為了評價H-SRM方法的優勢,我們用二個不同實驗分別檢測了代表24種人類血清蛋白的50種肽,一個是傳統的SRM全掃描MS/MS方法檢測,每個SRM測試使用了152組傳輸(每種肽2~3種離子對,每種蛋白1~4種肽)。第二個實驗沒有采用數據關聯全掃描MS/MS,使用了318組SRM傳輸(5~7)(每種肽5~7種離子對,每種蛋白1~4種肽)。為評價H-SRM方法消除血清背景離子干擾的能力,此實驗采用了高分辨(0.2 FWHM)和單位分辨(0.7FWHM)兩種方式。
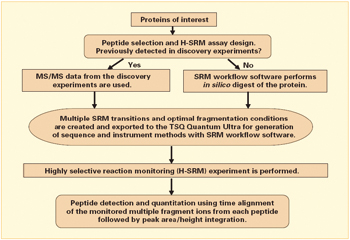
圖2. 同時進行肽檢測和定量的H-SRM工作流程。
TSQ Quantum Ultra質譜儀獲得的MS/MS數據分別使用SEQUEST?和Mascot在SwissProt數據庫進行了搜索,兩種搜索引擎給出了相似的結果。表1總結了Mascot得到的QED-MS/MS實驗數據的檢索結果。可以看出,從24種靶向蛋白中都確認出21種蛋白(補體成分1抑制劑、血漿激肽釋放酶和P<0.05的血紅蛋白α鏈沒有鑒定出來),TSQ Quantum Ultra質譜儀鑒定出36種肽。
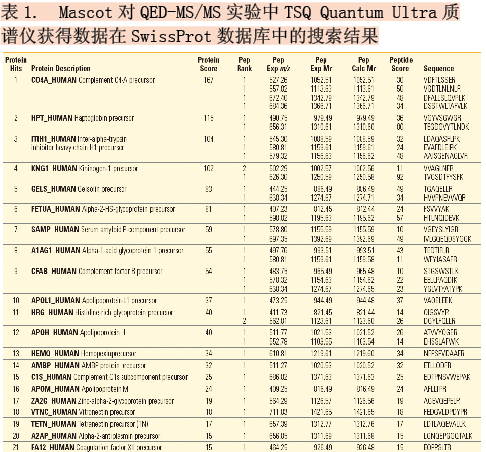
采用QED-MS/MS方法仍然沒有成功識別鑒定血清消化液中所有的50種目標肽。這主要是由于傳統方法在檢測復雜基體中的低豐度肽具有局限性,尤其是肽具有共流出的同量異位干擾物時,無法對其進行可靠鑒定2。從復雜的血清消化液中鑒定24種蛋白產生的50種目標肽的任務,只有用我們新提出的方法才可以完成,該方法采用高分辨分離來檢測低豐度肽,并用多碎片離子的時間序列來進行肽身份鑒定。圖3是一種低豐度肽(LLDSLPSDTR代表補體組分1抑制劑),傳統方法沒有將其鑒定出來,H-SRM方法則將其成功鑒定出來。H-SRM的TIC譜(上圖)信號峰信噪比與SRM的TIC譜(下圖)相比大約高12倍。母體通過5種y-離子的時間序列檢測得到了可靠的鑒定;Mascot序列搜索通過這些充分的數據搜索SwissProt數據庫,可以唯一地鑒定該肽。該方法無需全掃描MS/MS,具有分辨率分離能力,在低檢測限下快速可靠的鑒定目標肽方面具有很明顯的優勢。采用多碎片離子時間序列檢測的H-SRM方法鑒定了所有的50種肽。
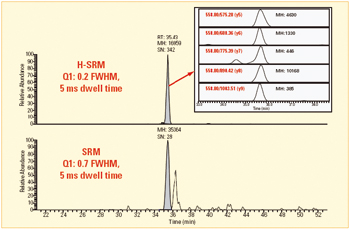
圖3. 通過多碎片離子時間序列檢測,H-SRM方法鑒定了代表補體組分1抑制劑蛋白的LLDSLPSDTR肽。與SRM方法相比,H-SRM方法顯著提高了信噪比,降低了血清基體非特異性干擾,可以明確地鑒定目標肽。
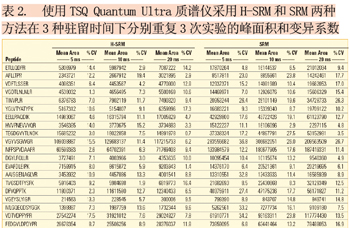
使用TSQ Quantum Ultra的H-SRM及SRM方法在不同駐留時間下測得的平均峰面積及變異系數(% CV)變化列于表2。H-SRM顯著降低了來自血清基體的非特異性干擾,在不同駐留時間下均獲得了較高的方法精密度,大約95%的肽的變異系數值小于15%。
定量準確度
H-SRM方法優良的分析精密度與其較高的定量準確度一致。H-SRM方法測定13種靶向蛋白的相對定量結果其平均誤差范圍為±4% (n=3)。
結論
本文介紹了TSQ Quantum
Ultra三級四極桿質譜儀采用H-SRM模式同時鑒定和定量靶向蛋白的新方法。該方法通過顯著降低血漿背景離子的非特異性干擾,在人類血清中靶向蛋白的分析中,顯示出良好的靈敏度、分析方法精密度和定量準確度。H-SRM方法極大地提高了方法專一性和檢測限,在生物標志物的驗證和確認研究中具有顯著優勢。
【參考文獻】
1. Leigh Anderson and Christie L. Hunter (2006) Quantitative Mass
Spectrometric Multiple Reaction Monitoring Assays for Major Plasma
Proteins. Mol. Cell. Proteomics 5 (4), 573-588.
2. Hasmik Keshishian, Terri Addona, Michael Burgess, Eric Kuhn and Steven A. Carr (2007) Quantitative, Multiplexed Assays for low Abundance Proteins in Plasma by Targeted Mass Spectrometry and Stable Isotope Dilution. MCP Papers in Press. Published on October 15, 2007 as manuscript M700354-MCP200.
3. Richard D. Unwin, John R. Griffiths, Michael K. Leverentz, Agnes Grallert, Iain M. Hagan and Anthony D. Whetton (2005) Multiple Reaction Monitoring to Identify Sites of Protein Phosphorylation with High Sensitivity. Mol. Cell. Proteomics 4 (8), 1134-1144.3.